
Vol. 39, n.º 3, 2006
REVISTA
ESPAÑOLA DE
Vol. 39, n.º 3, 2006 |
TÉCNICAS EN PATOLOGÍA
Patricia García M.1,2, Felipe Benavente M.1,2, Angélica Melo A.1, Iván Roa E.1,3, Juan Carlos Roa S.1
1 Laboratorio de
Patología Molecular, Dpto. Anatomía Patológica, Facultad de Medicina,
Universidad de La Frontera, Temuco, Chile.
2 Estudiante Doctorado de
biología molecular aplicada. Universidad de la Frontera.
3 Departamento de
Patología, Clínica Alemana, Santiago de Chile.
jcroa@ufro.cl
RESUMEN
Antecedentes: Los tejidos fijados y embebidos en parafina son una fuente importante de material para diagnóstico e investigación. La amplificación de ADN desde este tipo de tejidos, mediante reacción en cadena de la polimerasa (PCR), es afectada por el tipo de fijador y los tiempos de fijación empleados. Para determinar el parámetro que mejor se adecue a las condiciones de trabajo de nuestro laboratorio de Patología Molecular, evaluamos el efecto de cinco fijadores sobre la calidad del ADN bajo condiciones controladas. Material y Método: Muestras de mucosa gástrica fueron fijadas, embebidas en parafina y luego procesadas para extracción de ADN empleando un protocolo basado en digestión con proteinasa K. La calidad del ADN se evaluó mediante amplificación de tres fragmentos del gen b-globina (268, 536 y 989 pb). Resultados: No observamos mayores diferencias entre fijadores ni tiempos de fijación en la amplificación de ADN de 268 y 536 pb. No obstante, la amplificación del fragmento mayor (981 pb) se vio alterada al aumentar el tiempo de fijación, a excepción de aquellas muestras fijadas en etanol 70% que presentaron una banda de similar intensidad a la obtenida para muestras control (tejido fresco congelado). Conclusiones: Estos resultados nos proporcionan una pauta para el diseño de experimentos de acuerdo a la calidad del material archivado, optimizando recursos humanos e insumos.
Palabras clave: fijación, formalina, etanol 70%, reacción en cadena de la polimerasa, PCR.
SUMMARY
Background: Fixed and paraffin-embedded tissues from pathological archives are an important source for research. DNA amplification by polymerase chain reaction (PCR) from those materials is affected by fixatives and fixation time. In order to determine the best condition for our laboratory work, we evaluated the effect of five different fixatives on DNA quality under controlled conditions. Material and Method: Gastric mucosa samples from two patients were fixed, embedded in paraffin and then processed for DNA extraction using a routine method based on proteinase K digestion. DNA quality was evaluated by amplifying three b-globin gene fragments (268, 536 and 989 bp). Results: We did not observe major differences among fixatives nor fixation times for 268 and 536 bp fragments but the amplification of the greatest fragment was altered by increasing the fixation time, by excepting those samples fixed in 70% ethanol which present a similar band intensity than control samples (snap-frozen tissue). Conclusions: These results give us a good guideline to design experiments considering the quality of our archival material.
Key words: fixation, formalin, 70% ethanol, polymerase chain reaction, PCR.
INTRODUCCIÓN
Los tejidos fijados y embebidos en parafina constituyen un importante material de archivo para la realización de estudios clínicos retrospectivos y, además, representan un recurso invaluable para estudiar la patogenia molecular de las enfermedades y la expresión génica diferencial. La mayoría de los especimenes procesados en los laboratorios de Patología son rutinariamente fijados en formalina y, aunque esto permite conservar la arquitectura del tejido, la extracción de ADN y ARN de calidad adecuada para estudios de biología molecular es un problema significativo en la práctica de la Patología molecular (1-3).
Se ha demostrado que el formaldehído produce entrecruzamiento («cross-linkange») entre ácidos nucleicos y proteínas (4), hidroliza puentes fosfodiester del ADN (5) y modifica covalentemente el ARN por adición de grupos mono metilos en las bases (4,6), generando problemas en la obtención de material genético de buena calidad, necesario para la aplicación de variadas técnicas moleculares.
Al respecto, numerosos autores han demostrado que la fijación con formalina disminuye el rendimiento de la amplificación por PCR (reacción en cadena de la polimerasa), en mayor o menor grado dependiendo del tiempo y temperatura de fijación principalmente, factores que influirán en la tasa de fragmentación del ADN (7-9). Como un ambiente ácido es la principal causa de degradación, el uso de fijadores basados en formaldehído tamponado permite obtener mejores resultados (10). Otros estudios han evaluado el efecto de fijadores alternativos (etanol y derivados) que no produzcan «cross-linkage» y preserven en buen estado tanto la morfología del tejido como ácidos nucleicos y proteínas, de tal manera que el rendimiento del PCR sea superior (11-14). También se han evaluado y mejorado los métodos de extracción y desparafinación, demostrándose que el tratamiento con proteinasas mejora la recuperación de ADN durante la extracción, ya que permite liberar los ácidos nucleicos atrapados en complejos proteicos (15).
Considerando la información entregada, nos planteamos evaluar el efecto de cinco distintos fijadores y tiempos de fijación en la obtención de ADN óptimo para estudios moleculares, que además permitan mantener la morfología y preservar las estructuras proteicas de la muestra fijada, aspecto necesario para realizar los estudios rutinarios de inmunohistoquímica. De esta manera, buscamos establecer el mejor parámetro que se adecue a los procedimientos de rutina que se llevan a cabo en nuestro departamento de Anatomía Patológica.
MATERIAL Y MÉTODO
Muestras y fijación de los tejidos: Se utilizó fragmentos de mucosa gástrica no tumoral de dos pacientes, procesadas siguiendo el mismo protocolo. Los especimenes de la gastrectomía por cáncer fueron trasladados al laboratorio de patología dentro de los 30 minutos de la resección quirúrgica y pequeñas porciones de mucosa gástrica fueron distribuidas en 5 distintos fijadores: etanol 70%, formalina ácida 10% v/v, formalina buffer (formalina ácida 10% tamponada con buffer fosfatos, pH 7.0), Formal-Fixx (Shandon®, formalina tamponada libre de fosfatos) y fijador zinc (Tris 0,1 M pH 7,4; Acetato de calcio 3 mM; acetato de zinc 0,25 M; cloruro de zinc 0,4 M). Se evaluaron cuatro tiempos de fijación (6, 24, 72 y 168 horas) por cada fijador y, además, una porción de tejido se congeló inmediatamente en Nitrógeno líquido (–196°C) constituyendo nuestro gold standard.
Extracción de ADN genómico: Una porción del tejido fresco congelado fue transferida a 600 µl de buffer de lisis (10 mM Tris pH 7,8; 5 mM EDTA pH 8,0; 0,5% SDS) y tratado con proteinasa K (500 ug/ml). En el caso del tejido fijado y embebido en parafina, láminas de 10 µm fueron cortadas desde cada bloque, colocadas en portaobjetos y desparafinadas con 3 cambios de xilol y 3 de alcohol 100%, por 1 minuto cada vez. Este tejido fue microdisecado con una hoja de bisturí y transferido a tubos preparados con buffer de lisis y proteinasa K. Todas las muestras se incubaron a 65°C hasta la completa disolución del tejido y luego a 95°C por 10 minutos para inactivar la enzima. Para la precipitación de proteínas se empleó acetato de amonio 7,5 M pH 7,5. El ADN se precipitó con isopropanol, incubando a –20ºC toda la noche, se recuperó por centrifugación y se resuspendió en buffer TE pH 7,4. El ADN obtenido fue cuantificado, separado por electroforesis en gel de agarosa 1% y visualizado por tinción con bromuro de etidio.
Amplificación por PCR: La calidad del ADN total extraído se evaluó mediante amplificación por PCR de tres fragmentos del gen b-globina. La reacción de PCR se preparó con 150 ng de ADN como templado, 0,6 unidades de Taq ADN polimerasa (Promega), 1,5 mM MgCl2, 0,2 mM de cada dNTP y 0,45 uM de cada partidor. Los iniciadores empleados para amplificar los fragmentos de 268, 536 y 989 pb fueron respectivamente (en sentido 5’-3’): GH20 (GAAGAGCCAAGGACAGGTAC) y PCO4 (GGTGAACGTGGATGAAGTTG); RS42 (GGTTGGCCAATCTACTCCCAGG) y KM29 (CTTTGCCACACTGAGTGAGCT); RS80 (ATTTTCCCACCCTTAGGCTG) y RS40 (CAGCTACAATCCAGCTACCA). Las condiciones de PCR se optimizaron para cada par de iniciadores, variando las temperaturas de hibridación (50, 60 y 65°C) en cada caso. Los productos de PCR fueron separados en geles de agarosa al 2% y visualizados por tinción con bromuro de etidio.
RESULTADOS
La extracción de ADN empleando un protocolo basado en digestión con proteinasa K permitió obtener material suficiente para la amplificación mediante PCR. La proporción OD260/OD280 fue en promedio de 1,6 para las muestras fijadas y 1,75 para la muestra fresca congelada, indicando la obtención de ADN relativamente puro en ambos casos. Sin embargo, al visualizar el ADN por electroforesis en gel de agarosa es evidente el estado de fragmentación del ADN en los tejidos fijados y embebidos, no así cuando se extrajo desde el tejido fresco congelado con el cual se obtiene una banda de alto peso molecular. Se observa mayor deterioro del ADN al utilizar formalina ácida (FA) en la fijación de las muestras, no recuperándose ADN de alto peso molecular luego de 72 horas de fijación (fig. 1A).

Fig. 1: (A)
Calidad del DNA extraído desde los tejidos
fijados en comparación al control de tejido fresco congelado (2 ug DNA genómico
por carril). Las flechas (¤) indican la mayor
degradación del DNA en muestras fijadas en FA, no observándose DNA de alto peso
molecular en los pocillos de carga. Las flechas (Ø)
señalan la mayor fragmentación del DNA en las mismas muestras. (B, C, D)
Amplificación de tres fragmentos del gen b-globina
a partir de tejidos fijados y embebidos en parafina (268, 536 y 989 pb
respectivamente). En D se observa la disminución de la señal amplificada al
aumentar el tiempo de fijación para las muestras fijadas en FA, FB y FF. MP=marcador
de peso molecular. ET=Etanol 70%. FA=Formalina ácida. FB=Formalina
tamponada con fosfatos. FF=Formal-Fixx. FZ=Fijador Zinc. +=Control
(tejido fresco congelado). b=blanco.
Al evaluar la calidad del ADN extraído de servir como templado para la amplificación de distintos tamaños de fragmentos, empleando partidores específicos dirigidos al gen b-globina, fue posible amplificar los fragmentos de 268 y 536 pb para todos los fijadores y tiempos de fijación evaluados (fig. 1B y 1C), presentándose una disminución de la señal amplificada en las muestras fijadas durante 168 horas en FA. La tabla 1 resume los resultados del PCR de acuerdo a la intensidad de las bandas obtenidas, estimada por comparación con las muestras control (tejido fresco congelado).
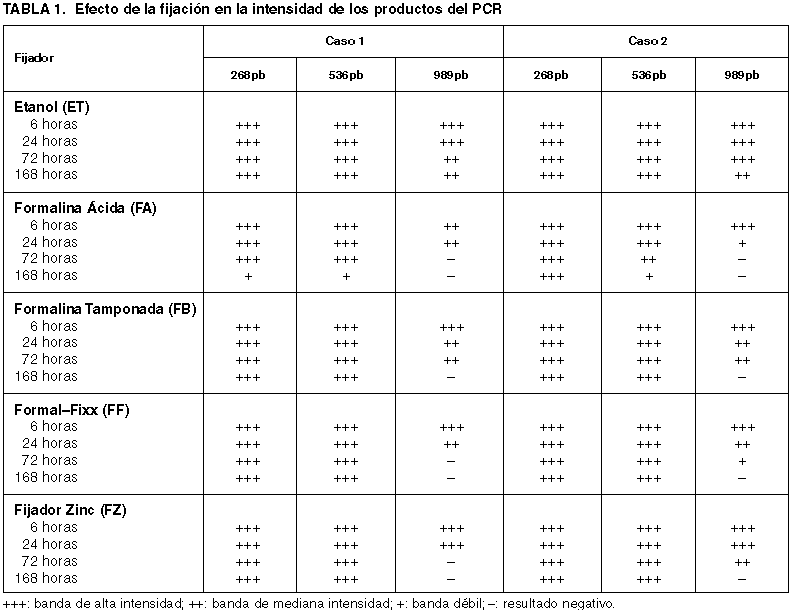
La amplificación de un fragmento mayor (989 pb) no mostró variación significativa entre fijadores a las 6 horas, presentando todas las muestras una intensidad de banda similar al control positivo de tejido congelado (fig. 1D). Desde las 24 horas de fijación comienza a disminuir el rendimiento del PCR para las muestras fijadas en FA, formalina buffer (FB) y Formal-Fixx (FF), obteniéndose bandas más tenues. A las 72 horas se repite este patrón para las muestras fijadas en FB y FF, pero ya no se observa amplificación del gen para el ADN extraído desde las muestras fijadas en FA. En el caso particular de las muestras fijadas en etanol (ET) y fijador zinc (FZ), hasta las 72 horas no hay variación con respecto al control en fresco. Transcurridas 168 horas de fijación, la señal para las muestras fijadas en FB, FF y FZ disminuye, conservándose todavía intensa para las muestras fijadas en etanol.
DISCUSIÓN
Los tipos de fijadores y tiempos de fijación influyen en la preservación de proteínas para inmunohistoquímica y también en los resultados del PCR (7,8). Los fijadores basados en formaldehído producen un gran entrecruzamiento de ácidos nucleicos y proteínas (3) lo cual obstaculiza la reacción de PCR. El etanol y otros fijadores basados en alcoholes no causan este entrecruzamiento y por lo tanto preservan adecuadamente los ácidos nucleicos para una posterior amplificación de ADN. Varios autores han observado que el etanol da los mejores resultados cuando se amplifican secuencias de genes a partir de material fijado y embebido en parafina, a diferencia de aquellos tejidos fijados en formalina que permiten recuperar ADN de baja calidad con el cual no es posible amplificar fragmentos muy grandes (7,11-13).
Nuestros resultados concuerdan con lo anterior, ya que desde las muestras de tejido fijadas con etanol 70% se obtuvo ADN de calidad suficiente para servir como templado en la amplificación de un fragmento de 989 pb del gen b-globina, desde 6 a 168 horas de fijación, con similar rendimiento al control de tejido congelado. Los demás fijadores, a excepción del fijador alternativo basado en zinc (FZ), permitieron amplificar el mismo tamaño de fragmento a las 6 horas de fijación, existiendo un deterioro o pérdida de la señal amplificada al aumentar el tiempo, lo cual ya ha sido observado por otros autores al evaluar tejido fijado en formalina (7,8). En el caso particular de las muestras fijadas en FZ, la señal se mantuvo sin variación hasta las 72 horas, pero a las 168 horas el rendimiento decayó fuertemente. Al respecto, ya se ha reportado un mejor rendimiento en la obtención de ADN al utilizar este tipo de fijador, en comparación a tejidos fijados en formalina tamponada neutra (16). Además, en el mismo estudio, determinan que la inmunoreactividad fue superior después de la fijación con este reactivo, al compararla con tejidos fijados en formalina, aunque disminuye levemente la calidad morfológica (16). No existen reportes acerca de la influencia del tiempo de fijación sobre la calidad del ADN obtenido a partir de muestras tratadas con fijadores basados en zinc.
En cuanto a los fijadores tamponados, no encontramos diferencias concluyentes entre la formalina tamponada con buffer fosfatos y la comercial Formal-Fixx (libre de fosfatos). El rendimiento del PCR para el fragmento de 989 pb fue disminuyendo en ambos casos a partir de las 24 horas evaluadas, hasta ser apenas perceptible a las 168 horas.
Nuestro trabajo concuerda ampliamente con otros resultados publicados, en cuanto a que el tratamiento de muestras con formalina ácida contribuye a un menor rendimiento de ADN óptimo para ser empleado en amplificación mediante PCR y que la fijación con etanol 70% entrega los mejores resultados. Sin embargo, cabe señalar que nosotros logramos amplificar fragmentos de hasta 500 pb, sin diferencias entre fijadores (incluida la formalina ácida) y sin variación de la señal amplificada al aumentar el tiempo de fijación hasta 168 horas. Otros trabajos han reportado amplificaciones no mayores a 420 pb y sólo hasta 8 horas de fijación en muestras fijadas en formalina ácida (8,11).
La información obtenida es importante para el funcionamiento de nuestro laboratorio, ya que con anterioridad al año 1998 gran parte de nuestro material fue fijado en formalina ácida por tiempos variables. Este hecho nos conduce a racionalizar los recursos humanos e insumos de nuestro laboratorio con experimentos diseñados de manera adecuada considerando la calidad del material archivado.
BIBLIOGRAFÍA
Tokuda Y, Nakamura T, Satonaka K, Maeda S, Doi K, Baba S, et al. Fundamental study on the mechanism of DNA degradation in tissues fixed formaldehyde. J Clin Pathol 1990; 43: 748-51.
Yagi N, Satonaka K, Horio M, Shimogaki H, Tokuda Y, Maeda S. The role of DNase and EDTA on DNA degradation in formaldehyde fixed tissues. Biotech Histochem 1996; 71: 123-9.
Srinivasan M, Sedmak D, Jewell S. Effect of fixatives and tissue processing on the content and integrity nucleic acids. Am J Pathol 2002; 161: 1961-71.
Feldman MY. Reactions of nucleic acids and nucleoproteins with formaldehyde. Prog Nucleic Acid Res Mol Biol 1973; 13: 1-49.
Douglas MP, Rogers SO. DNA damage caused by common cytological fixatives. Mutat Res 1998; 401: 77-88.
Masuda N, Ohnishi T, Kawamoto S, Monden M, Okubo K. Analysis of chemical modification of RNA from formalin-fixed samples optimization of molecular biology applications for such samples. Nucleic Acids Res 1999; 27: 4436-43.
Ben-Ezra J, Johnson DA, Rossi J, Cook N, Wu A. Effect of fixation on the amplification of nucleic acids from paraffin-embedded material by the polymerase chain reaction. J Histochem Cytochem 1991; 39: 351-4.
Karlsen F, Kalantari M, Chitemerere M, Johansson B, Hagmar B. Modifications of human and viral deoxyribonucleic acid by formaldehyde fixation. Lab Invest 1994; 71: 604-11.
Romero RL, Juston AC, Ballantyne J, Henry BE. The applicability of formalin-fixed and formalin fixed paraffin tissues in forensic DNA analysis. J Forensic Sci 1997; 42: 708-14.
Zsikla V, Baumann M, Cathomas G. Effect of buffered formalin on amplification of DNA from paraffin wax embedded small biopsies using real-time PCR. J Clin Pathol 2004; 57: 654-6.
Giannella C, Zito FA, Colonna F, Paradiso A, Marzullo F, Alaibac M, et al. Comparison of formalin, ethanol, and Histochoice fixation on the PCR amplification from paraffin-embedded breast cancer tissue. Eur J Clin Chem Clin Biochem 1997; 35: 633-5.
Gillespie JW, Best CJ, Bichsel VE, Cole KA, Greenhut SF, Hewitt SM, et al. Evaluation of non-formalin tissue fixation for molecular profiling studies. Am J Pathol 2002; 160: 449-57.
Alaibac M, Filotico R, Giannella C, Paradiso A, Labriola A, Marzullo F. The effect of fixation type on DNA extracted from paraffin-embedded for PCR studies in dermatopathology. Dermatology 1997; 195: 105-7.
Noguchi M, Furuya S, Takeuchi T, Hirohashi S. Modified formalin and methanol fixation methods for molecular and morphological analyses. Pathol Int 1997; 47: 685-91.
Diaz-Cano SJ, Brady SP. DNA extraction from formalin-fixed, paraffin-embedded tissues: protein digestion as a limiting step for retrieval of high-quality DNA. Diagn Mol Pathol 1997; 6: 342-6.
Wester K, Asplund A, Backvall H, Micke P, Derveniece A, Hartmane I, et al. Zinc-based fixative improves preservation of genomic DNA and proteins histoprocessing of human tissues. Lab Invest 2003; 83: 889-99.