
Vol. 39, n.º 3, 2006
REVISTA
ESPAÑOLA DE
Vol. 39, n.º 3, 2006 |
TÉCNICAS EN PATOLOGÍA
Yaxsier de Armas1, Virginia Capó1, Efraín González1, Lilian Mederos2, Raúl Díaz2
1 Departamento de
Anatomía Patológica del Instituto de Medicina Tropical «Pedro Kourl».
2 Laboratorio Nacional de Referencia e Investigaciones de
Tuberculosis y Micobacterias del Instituto de Medicina Tropical «Pedro Kourl».
yaxsier@ipk.sld.cu
RESUMEN
Antecedentes: El tejido embebido en parafina (TEP) es una fuente invaluable para los estudios moleculares retrospectivos. El método de Chelex-100 ha sido empleado para la extracción de ADN en TEP con el objetivo de ser utilizado posteriormente en la técnica de la Reacción en Cadena de la Polimerasa (RCP). En este trabajo, se describe la utilidad del Chelex-100 para la detección de ADN de Mycobacterium tuberculosis de TEP. Métodos: Se realizaron las técnicas de Hematoxilina-Eosina y Ziehl-Neelsen a TEP de pacientes fallecidos por SIDA. Adicionalmente, se utilizó el método de Chelex-100 para extraer ADN y evaluar dos juegos de iniciadores para la detección de M. tuberculosis empleando la RCP. Resultados: Con el empleo del Chelex-100 se obtuvo un material genético con buena calidad y cantidad suficiente. Se identificó M. tuberculosis en las muestras analizas a través de la amplificación de la secuencia específica de inserción (IS6110), demostrando la utilidad de la resina quelante para la RCP de fragmentos cortos. Conclusión: El método del Chelex-100 brinda una opción menos costosa, rápida, fácil y segura para los estudios moleculares en Anatomía Patológica.
Palabras claves: extracción ADN; Chelex-100; parafina; RCP; mycobacterium tuberculosis.
SUMMARY
Introduction: Paraffin-embedded tissue (PET) is an invaluable resource for retrospective molecular studies. Chelex-100 method has been used to DNA extraction from PET. The DNA obtained is used in Polymerase Chain Reaction (PCR) technique. In this work, the utility of Chelex-100 method for the detection of Mycobacterium tuberculosis is showed. Methods: The Hematoxilyn-Eosin and Ziehl-Neelsen techniques were performed in PET samples from three patients dying of AIDS. Additionally, Chelex-100 method was used for DNA extraction from these tissues and two PCR with different primers were evaluated for identification of M tuberculosis. Results: Genetic material with a good quality and in enough quantity was obtained the Chelex-100 method. M tuberculosis was identified in the samples by PCR, using a specific sequence (IS 6110). We showed the utility of Chelex-100 method in PCR from short DNA fragments. Conclusions: The Chelex-100 method is a rapid, easy, safe and inexpensive option in molecular Pathology research.
Key words: DNA extraction; Chelex-100; paraffin; PCR; mycobacterium tuberculosis.
INTRODUCCIÓN
El análisis de los ácidos nucleicos se ha convertido en una herramienta importante en el diagnóstico e investigación en patología molecular. El material más comúnmente analizado en los laboratorios de esta disciplina es el tejido embebido en parafina (TEP) (1). El material biológico conservado de esta forma constituye una fuente incalculable de información en investigaciones de carácter retrospectivo. Sin embargo, la obtención de ADN genómico de alta calidad a partir de TEP es una tarea difícil (2).
En la última década se han publicado varios métodos para la extracción y obtención de ADN a partir de TEP. Algunos protocolos, por ejemplo, utilizan diferentes tiempos de incubación para el tratamiento proteolítico de las muestras (3), otros emplean solventes orgánicos adicionales como fenol y cloroformo, antes de la precipitación con etanol (4). Existen metodologías alternativas, que utilizan la lisis celular por calentamiento en agua destilada para procesar el tejido (5). Por otra parte, en la literatura internacional han aparecido protocolos que emplean la sonicación de la muestra y la incubación de la misma en una solución con varios detergentes (6). Recientemente, otros autores han aplicado sistemas comerciales para la purificación de ADN en TEP (7).
El empleo del Chelex-100 en el campo de la genética molecular también ha sido explorado. Varios grupos de investigación han utilizado esta resina quelante para obtener ADN en bloques de parafina: en las ciencias forenses (8), en la detección molecular de diferentes especies de bacterias (9) y en estudios virológicos (10). En el presente trabajo, se empleó Chelex-100 para la extracción de ADN de Mycobacterium tuberculosis de TEP.
MATERIALES Y MÉTODOS
Muestras clínicas
Se utilizó material de tejido embebido en parafina de tres pacientes con el Síndrome de Inmunodeficiencia Adquirida (SIDA), fallecidos en el Instituto de Medicina Tropical «Pedro Kourí» (IPK), Ciudad de la Habana, Cuba. Paralelamente, se tomó una muestra similar de cada fallecido para la confirmación diagnóstica por la técnica de cultivo en el Laboratorio Nacional de Referencia e Investigaciones de Tuberculosis y Micobacterias del IPK.
Procesamiento del tejido
Se realizó un corte histológico (de cada bloque) de 5 µm en un micrótomo RM 2035 (Leica, Alemania) y se coloreó con hematoxilina y eosina para dar un diagnóstico presuntivo de infección por micobacterias. Se tomó otro corte para realizar la coloración de Ziehl-Neelsen, específica para micobacterias (11).
Se utilizó además un corte de 14 µm del tejido de cada bloque de parafina y se colocó en un microtubo de 0,5 mL para realizar la Reacción en Cadena de la Polimerasa (RCP).
Extracción de ADN
El procedimiento de extracción de ADN del corte histológico del bloque de parafina fue llevado a cabo utilizando 150 µL de una solución de Chelex-100 (Sigma-Aldrich, USA) al 5% y calentando a 100°C por 30 minutos. Luego se centrifugó a 13.000 rpm por 10 min y se transfirió el sobrenadante a un microtubo de 0,5 mL estéril, sin tomar la resina quelante para evitar inhibición de la RCP (12).
Reacción en Cadena de la Polimerasa (RCP)
Se tomaron múltiples precauciones para evitar cualquier tipo de contaminación en las muestras analizadas, a partir de lo reportado por Kwok (13).
Se utilizó como secuencia diana un fragmento de 245 pares de bases (pb) de la secuencia de inserción 6110 (IS6110) de M. tuberculosis (14) y otro de 439 pb contenido en la secuencia nucleotídica de la proteína de 65 KDa de micobacterias (15). La mezcla de reacción (50 µL) para el fragmento de 245 pb contuvo Tris/HCl (pH 8,3) 10 mM, KCl 50 mM, MgCl2 1,5 mM, deoxirribonucleótidos (dNTP) 200 µM, iniciador (INS-1 e INS-2) 0,4 µM, 2 unidades de Taq ADN polimerasa (Bioline, Reino Unido), y 25 µL de ADN molde. El perfil de amplificación fue: 96°C por 3 min., 30 ciclos de 1 min. a 96°C, 1 min. a 65°C y 2 min. a 72°C, con una extensión final de 6 min. a 72°C.
Para el fragmento de 439 pb la mezcla de reacción (50 µL) incluyó Tris/HCl (pH 8,3) 10 mM, KCl 50 mM, MgCl2 1,5 mM, dNTPs 200 µM, iniciador (TB11 y TB12) 0,5 µM, 1,25 unidades de Taq ADN polimerasa (Bioline, Reino Unido), y 25 µL de ADN molde. El perfil de RCP fue: 96°C por 3 min., 35 ciclos de 1 min. a 94°C, 1 min. a 60°C y 1 min. a 72°C, con una extensión final de 6 min. a 72°C. Todas las amplificaciones se realizaron en un termociclador Minicycler PTC 150 (MJ. Research, EUA).
Para la detección de los productos amplificados se analizaron 20 µL de cada mezcla resultante en una electroforesis en gel de agarosa al 1,2% con solución reguladora de Tris-borato-EDTA. La corrida se realizó a 80 Volts por 1 hora. Los resultados fueron visualizados en un transiluminador ultravioleta (Macrovue 2011, LKB, Suecia) y luego fotografiados con una cámara digital Power Shot G6 (Cannon, Japón).
En todas las reacciones de RCP se empleó ADN extraído del cultivo de una cepa de M. tuberculosis (H37RV) gentilmente entregada por el Laboratorio Nacional de Referencia e Investigaciones de Tuberculosis y Micobacterias, como control positivo y agua destilada estéril (en lugar de ADN molde) como control negativo.
RESULTADOS
El Chelex-100 es una resina que preferentemente captura iones metálicos polivalentes en soluciones. El efecto de esta sustancia es prevenir a través de su acción quelante la ruptura del ADN a altas temperaturas catalizada por los iones involucrados en las muestras de trabajo.
En esta investigación, con el empleo del Chelex-100 se obtuvo un material genético con buena calidad y en cantidad suficiente para detectar M. tuberculosis, de los tres bloques de TEP analizados, empleando la RCP.
En la figura 1 se pueden observar las bandas de 245 pb amplificadas obtenidas a partir de los ADN extraídos de cada bloque de parafina, preparado con una muestra de cada paciente estudiado.
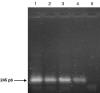
Fig 1:
RCP (IS6110) específico para el complejo
Mycobacterium tuberculosis 1: Control positivo (ADN de M. tuberculosis [H37RV]);
2: Muestra 1; 3: Muestra 2; 4: Muestra 3; 5: Control negativo de la reacción.
Al realizar la RCP, a los tres bloques de TEP, con el fragmento de 439 pb correspondiente a una región conservada de la proteína 65 KDa para todas las micobacterias, no se observó ningún producto de amplificación en el gel de agarosa.
DISCUSIÓN
El fragmento de inserción IS6110 se ha convertido en una secuencia diana muy empleada para la detección de M tuberculosis en TEP, debido a su especificidad y repetibilidad en el genoma de la bacteria (16).
Diferentes autores han planteado que la amplificación de fragmentos de ADN en TEP dependen de varios factores: tamaño del fragmento diana a amplificar, copias del fragmento en el genoma, concentración de ADN, fijación de la muestra, entre otros (17,18). En nuestros resultados, todas las muestras fueron fijadas de igual manera, con formalina al 10%. Las concentraciones de ADN fueron chequeadas previamente por electroforesis y no existieron diferencias entre ellas. Sin embargo, parece ser que influye directamente el número de copias del fragmento en el genoma, pues el gen que codifica para la proteína 65 KDa está presente en una única copia en el genoma. Por otra parte, el fragmento diana a amplificar es de mediano peso molecular (439 pb), lo que pudiera limitar la obtención del producto de la RCP (17).
A pesar de este inconveniente, la extracción de ADN de TEP por Chelex-100 permitió la obtención rápida de ADN de M tuberculosis con un mínimo de manipulación y por lo tanto, de reducidas posibilidades de introducción de contaminantes en el proceso de purificación.
La extracción de ADN de tejidos embebidos en parafina por la resina quelante (Chelex-100) es menos costosa que otros métodos que emplean reactivos caros como la proteinasa K y los sistemas comerciales. Es también una metodología segura, pues no afecta la salud como el método que emplea el fenol (19). Podemos destacar además, que es un protocolo muy simple pues en un único paso se obtiene ADN con calidad suficiente para la técnica de la RCP y sólo se necesitan dos microtubos para su realización.
El método del Chelex-100 brinda una opción rápida y menos costosa para obtener ADN de TEP, para amplificar fragmentos diana de bajo peso molecular, constituyendo una opción ventajosa para los estudios moleculares en Anatomía Patológica.
BIBLIOGRAFÍA
Wiedorn KH, Olert J, Stacy R, Goldmann T, Kühl H, Matthus J. HOPE. A New Fixing Technique Enables Preservation and Extraction of High Molecular Weight DNA and RNA of >20 kb from Paraffin-embedded Tissues. Pathol Res Pract 2002; 198: 735-40.
Wu L, Patten N, Yamashiro CT, Chui B. Extraction and Amplification of DNA from Formalin-Fixed, Paraffin-Embedded Tissues. Applied Immunohistochem Mol Morphol 2002; 10: 269-74.
Jackson DP, Lewis FA, Taylor GR, Boylston AW, Quirke P. Tissue extraction of DNA and RNA and analysis by the polymerase chain reaction. J Clin Pathol 1990; 43: 499-504.
Wright DK, Manos MM. Sample preparation from paraffin-embedded tissues. En: Innis MA, editor. PCR protocols: a guide to methods and applications. San Diego: Academic Press; 1990. p. 153-6.
Lench N, Stainer P, Williamson R. Simple non-invasive method to obtain DNA for gene analysis. Lancet 1988; 8599: 1356-8.
Heller MJ, Robinson RA, Burgart U, TenEyck CJ, Wilke WW. DNA extraction by sonication: a comparison of fresh, frozen, and paraffin-embedded tissues extracted for use in polymerase chain reaction assays. Mod Pathol 1992; 5: 203-6.
Hellmann A, Rohleder U, Schmitter H, Wittig M. STR typing of human telogen hairs-A new approach. Int J Legal Med 2001; 114: 269-73.
Romero RL, Juston AC, Ballantyne J, Henry BE. The applicability of formalin-fixed and formalin fixed paraffin embedded tissues in forensic DNA analysis. J Forensic Sci 1997; 42: 708-14.
Stein A, Raoult D. A simple method for amplification of DNA from paraffin embedded tissues. Nucleic Acid Res 1992; 20: 5237-8.
Pak F, Pyakural P, Kokhaei P, Kaaya E, Pourfathollah AA, Selivanova G, et al. HHV-8/KSHV during the development of Kaposi's sarcoma: evaluation by polymerase chain reaction and immunohistochemistry. Cutan Pathol 2005; 32: 21-7.
Luna LG, editors. Manual of histologic staining methods of the Armed Forces Institute of Pathology, 3ra ed. New York: McGraw-Hill,Inc; 1967.
Van der Zanden AGM, Hoentjen AH, Heilmann FGC, Weltevreden EF, Schouls LM, van Embden JA. Simultaneous detection and strain differentiation of Mycobacterium tuberculosis complex in paraffin wax embedded tissues and in stained microscopic preparations. J Clin Pathol 1998; 51: 209-14.
Kwok S, Higuchi R. Avoiding false positives with PCR. Nature 1989; 339: 237-8.
van Embden JDA, Cave MD, Crawford JT, Dale JW, Eisenach KD, Gicquel B. et al. Strain identification of Mycobacterium tuberculosis by DNA fingerprinting: recommendations for a standardized methodology. J Clin Microbiol 1993; 31: 406-9.
Telenti A, Marchesi F, Balz M, Bally F, Bottger EC, Bodmer T.Rapid identification of mycobacteria to the species level by polymerase chain reaction and restriction enzyme analysis. J Clin Microbiol 1993; 31: 175-8.
Zink AR, Nerlich AG. Molecular strain identification of the Mycobacterium tuberculosis complex in archival tissue samples tuberculosis. J Clin Pathol 2004; 57: 1185-92.
Marchetti G, Gori A, Catozzi L, Vago L, Nebuloni M, Rossi MC, et al. Evaluation of PCR in detection of Mycobacterium tuberculosis form formalin-fixed, paraffin-embedded tissues: comparison of four amplification assays. J Clin Microbiol 1998; 36: 1512-7.
Pamikar N, Siddiqi N, Dev G, Sharma S, Madan N, Hasnain SE. Comparison of polymerase chain reaction with conventional techniques for the detection of Mycobacterium tuberculosis from paraffin-embedded tissue sections in extrapulmonary tuberculosis. J Infect Dis Antimicrob Agents 2003; 20: 61-8.
Reneike A, Karlovsky P, Zebitz CPW. Preparation and purification of DNA from insect for AFLP analysis. Insect Mol Biol 1998; 7: 95-9.