
Vol. 39, n.º 1, 2006
REVISTA
ESPAÑOLA DE
Vol. 39, n.º 1, 2006 |
ORIGINALES
Juliana Escobar Stein1, Aurora Astudillo González2, Primitiva Menéndez Rodríguez3, Elena Belyakova4
1 Servicio de
Patología, Hospital de Jarrio (Asturias).
2 Banco de tumores del Hospital Universitario Central de
Asturias-IUOPA.
3 Servicio de Patología, Hospital Monte Naranco (Oviedo).
4 Bioestadística del Instituto Universitario de Oncología del
Principado de Asturias (Oviedo).
julis74@hotmail.com
RESUMEN
Los tissue microarrays (TMAs) hacen posible agrupar más de 1.000 tumores en una sola laminilla, y ser utilizados para el análisis de numerosos marcadores de interés diagnóstico, pronóstico o de ayuda en la decisión terapéutica y selección de estudios moleculares. Así hace posible la detección y estudio de DNA y RNA (por hibridación in situ o FISH) y proteínas (inmunohistoquímica).
El objetivo de este estudio fue comprobar la representatividad de los TMAs en el estudio morfológico de diferentes tumores y valorar los TMAs como herramienta para el estudio inmunohistoquímico. Los TMA se realizaron en adenocarcinomas de pulmón, adenocarcinomas de colon, carcinomas escamosos y oat-cell pulmonares, en el Hospital Universitario Central de Asturias. Pudimos comprobar que los TMAs con tres cilindros por cada caso, son representativos del tumor, tanto cuantitativamente como cualitativamente.
La utilización de esta técnica tiene grandes ventajas que hacen posible efectuar un estudio en un tiempo escaso. Ello se halla motivado por una rápida velocidad en el procesamiento de las muestras, la posibilidad de análisis simultáneo y estandarizado de múltiples muestras y la existencia de un área de tamaño suficiente para analizar.
Palabras clave: tissue microarrays, inmunohistoquimica, EGFR, Ki-67.
SUMMARY
Tissue microarrays (TMAs) allow the congregation of more than 1000 tumors in a single microscope slide. It can be used for diagnostic and/or prognostic biomarker analysis that permits the evaluation of therapeutic strategies or selection of molecular studies. The TMAs allow the detection of DNA and RNA by fluorescent in situ hybridization and protein detection by immunohistochemistry.
The purpose of this study was to evaluate the ability of TMAs to differentiate tumors in morphologic studies and to assess the potential of the TMAs as a tool for immunohistochemistry. The TMAs were performed in lung adenocarcinoma, colon adenocarcinoma, squamous carcinomas and lung oat-cell carcinoma. The study was carry out on the Hospital Universitario Central de Asturias in Spain. We found that 3 cylinder cuts of each sample provided a quantitative and qualitative representation of each tumor. The quick processing time of the samples, the ability to analyze multiple samples at the same time, and wide area for analysis are the advantages of this technique. In conclusion the TMAs is a promising tool for investigation that will help in getting results in a shorter period of time.
Key words: tissue microarrays, immunohistochemistry, EGFR, Ki-67.
INTRODUCCIÓN
Muchos de los genes y señales que controlan la proliferación, muerte y diferenciación celular, así como la integridad genómica, están involucrados en el desarrollo del cáncer (1). El estudio de estos genes es complejo ya que los tejidos malignos pueden manifestarse con diversas alteraciones moleculares, como es la variabilidad dentro del mismo tumor o la sobreexpresión de oncogenes. Es importante estudiar estos cambios para hallar marcadores que ayuden en el diagnóstico, tratamiento y pronóstico de los tumores (2,3).
Así mismo, el avance en los estudios genómicos y proteonómicos están aumentando las necesidades de evaluar gran número de marcadores de utilidad diagnóstica, y que tengan un valor predictivo o pronóstico (4).
En los últimos años se han descrito nuevas técnicas de análisis genético que permiten valorar los genes implicados en el desarrollo de una enfermedad.
Las técnicas convencionales de patología molecular son complejas, requieren mucho tiempo y mucho tejido, limitando así, el número de tejidos y el número de marcadores que pueden ser evaluados. Por lo tanto se han desarrollado métodos menos complejos y que requieren menos tiempo (5). Entre los que encontramos los cDNA microarrays (micromatrices de cDNA) y los «tissue microarrays» (TMAs), o micromatrices de tejido. La técnica de cDNA microarrays permite estudiar los cambios en la expresión de un gran número de genes en una sola muestra tumoral (6), y los tissue microarrays permiten valorar las alteraciones genéticas bien a nivel del DNA, RNA o en las proteínas por ellos producidas en series amplias de un mismo tumor (7,8).
Los tissue microarrays (TMAs) hacen posible agrupar más de 1.000 tumores en una sola laminilla, y ser utilizados para el análisis de numeroso marcadores moleculares, así como para la detección y estudio de forma paralela de DNA (por hibridación in situ o FISH), RNA y proteínas diana (inmunohistoquímica). Permitiendo el análisis de un gran número de marcadores moleculares (9-11).
En Anatomía Patológica los TMAs también pueden tener muchas aplicaciones, ya que sirven como un instrumento de control de calidad para evaluar la sensibilidad y especificidad de los anticuerpos, métodos de fijación de los tejidos y optimizar protocolos de tinción, además pueden ayudar a optimizar y estandarizar la interpretación de la inmunohistoquímica (12).
Otro posible uso futuro de los Tissue Microarrays es la provisión de muestras randomizadas representativas de una lesión, susceptibles de ser evaluadas por métodos automáticos (reconocimiento de imagen: densidad nuclear, densidad cromática, resultados de tinciones inmunohistoquímicas) para abrir camino a un diagnóstico objetivo automatizable en Anatomía Patológica, que es, hoy por hoy, uno de los laboratorios diagnósticos con más intervención humana «ars medica» y mayor subjetividad diagnóstica (13,14).
Una de las posibles desventajas de esta nueva técnica es el reducido tamaño de la muestra de tumor, ya que se utilizan cilindros de 0,6 mm, lo que representa aproximadamente sólo el 0,3% del tumor (15), por lo cual se han realizado varios estudios de validación de la técnica (16-18).
En el año 2000, Gillett y cols.(19) efectúan un estudio de la expresión de receptores de estrógenos (ER) y progesterona (PR) en carcinomas de mama comparando su expresión respecto a la de tejidos en bloques enteros y la expresión obtenida de tres cilindros. Se demuestra una alta representatividad de los marcadores inmunohistoquímicos en los cilindros del TMAs. Las mayores discrepancias se encontraron en los tumores más heterogéneos.
Ese mismo año, Camp y cols. (15) realizan un estudio también en carcinomas de mama evaluando la expresión de ER, PR y Her2/Neu, para valorar cuantos cilindros eran representativos y evalúan como influye el paso del tiempo en la expresión de los marcadores. Encuentran que el porcentaje de positividad era acorde en los cilindros y las secciones completas, no encuentran cambios significativos en la positividad de tinciones antiguas.
El objetivo de este estudio es comprobar la representatividad de los TMAs en el estudio morfológico de diferentes tumores y valorar los TMAs como herramienta para el estudio inmunohistoquímico.
MATERIALES Y MÉTODOS
Los TMA se realizaron en 18 Adenocarcinomas de Pulmón, 18 Adenocarcinomas de Colon, 18 Carcinomas Escamosos y 8 Oat-Cell pulmonares, diagnosticados en el período de 1998-2002 en el Hospital Universitario Central de Asturias (HUCA).
El estudio consistió en efectuar tinciones simples de hematoxilina-eosina e inmunohistoquímica sobre los cortes completos de las muestras representativas del tumor, repitiendo el mismo estudio en los Tissue Microarrays, obtenidos del mismo bloque.
Se diseñó un sistema manual para la extracción e inserción de los cilindros. La técnica consistió en tomar un pequeño cilindro de cada bloque de parafina (bloque donante) e insertarlo de forma precisa en un bloque receptor de parafina. Se utilizaron cilindros de 1 mm y se seleccionaron las zonas más representativas del tumor en los cortes de hematoxilina-eosina (fig. 1). De cada tipo histológico se construyeron los tissue microarrays con tres representaciones del tumor. Los cilindros fueron obtenidos del centro, periferia y zonas intermedias del tumor.
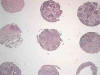
Fig. 1:
TMAs de carcinomas escamosos (HE x10).
La homogeneización de las muestras una vez introducidas en el bloque receptor se realizó mediante calor. Se anotó la posición de cada cilindro, para poder identificarlos posteriormente. Se realizaron cortes de 4 micras para la tinción con hematoxilina-eosina y estudio inmunohistoquímico.
Para el estudio inmunohistoquímico se utilizaron anticuerpos monoclonales tanto para el EGFR (DAKO clon H11, dilución 1:300) como para el Ki-67 (DAKO clon MIB-1, dilución 1:100). Se eligieron los citados anticuerpos para obtener resultados cualitativos (EGFR) y cuantitativos (Ki-67).
El Ki-67 requirió pretratamiento con alta temperatura (olla a presión por 30 minutos), mientras que para el EGFR se utilizó pretratamiento con proteinasa K por 15 minutos. Se utilizó en protocolo estándar de tinción.
La interpretación del EGFR se hizo usando un sistema de 4 puntos, según Goldstein (20):
0: No reactivo.
1+: Reacción débil: Tinción débil o incompleta del citoplasma (fig. 2).
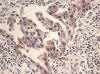
Fig. 2:
EGFR intensidad débil (1+) (HE x10).
2+: Reacción moderada: Intensidad moderada, completa o incompleta de la membrana (fig. 3).
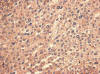
Fig. 3:
EGFR intensidad moderada (2+) (HE x10).
3+: Reacción intensa: Marrón oscuro, la mayoría de las veces completa de la membrana, produciendo engrosamiento de la célula (fig. 4).
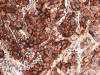
Fig. 4:
EGFR intensidad fuerte (3+) (HE x10).
4: No valorable/perdido.
Para la interpretación del Ki-67 se utilizó una escala de 4 niveles:
Índice de proliferación bajo: Menor del 15% de las células positivas.
Índice de proliferación medio: Entre 15-25% de las células positivas.
Índice de proliferación alto: Más del 25% de las células positivas.
Para la valoración cuantitativa del Ki-67 se emplea Imagen-Pro Plus versión 4.5.1. Para el análisis de la expresión del EGFR se utilizó una estimación subjetiva con doble observador.
ESTADÍSTICA
La meta fue comparar los resultados procedentes de las secciones completas del bloque tumoral con los TMA. El tamaño de la muestra, que consta de 62 tumores, era suficiente, la fiabilidad de los datos es media-alta. El estudio descriptivo incluía: la media, la desviación típica y los intervalos de confianza al 95% para las mediciones sucesivas efectuadas en las áreas de interés. En todo caso, la desviación típica no excedió la tercera parte de la media.
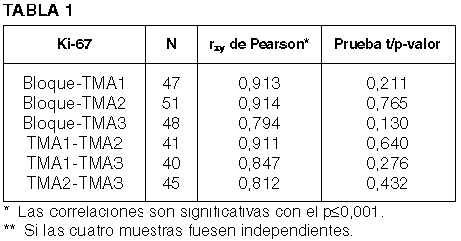
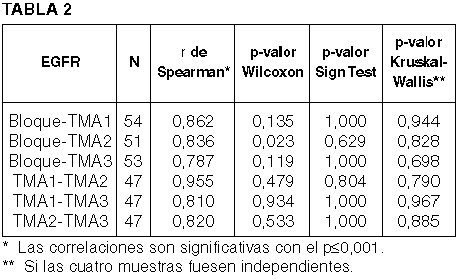
Para el análisis estadístico se han aplicado varios procedimientos como: las correlaciones de Pearson y de Spearman, las pruebas t de Student para las muestras relacionadas, el test de Wilcoxon, el test de signos y el test de Kruskal-Wallis.
Además en el Bloque se demostró la compatibilidad entre los marcadores Ki-67 y EGFR con el test de Wilcoxon de los rangos.
RESULTADOS
Ki-67: La prueba t para el Bloque y la muestra compuesta por los tres cilindros (La muestra compuesta de los TMA fue calculada como la suma de los recuentos de los tres cilindros dividida entre tres). TMA obtiene un p-valor=0,164 y el intervalo de confianza para la diferencia de las medias contiene el punto cero; y el coeficiente de correlación de Pearson entre ellos es de 0,920 (p–<0,01). En la muestra de 62 tumores, las correlaciones entre los valores del Bloque y de los TMA son altas y los p-valores de las pruebas t están muy por encima de 0,05 (figs. 5-12).

Fig. 5:
Positividad de Ki-67 en carcinoma escamoso en
bloque (HE x10).
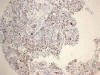
Fig. 6:
Positividad de Ki-67 en carcinoma escamoso en
TMAs (HE x10).
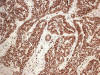
Fig. 7:
Positividad de Ki-67 en adenocarcinoma de colon
en bloque (HE x10).
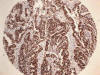
Fig. 8:
Positividad de Ki-67 en adenocarcinoma de colon
en TMAs (HE x10).
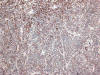
Fig. 9:
Positividad de Ki-67 en oat-cell en bloque (HE
x10).
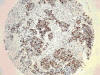
Fig. 10:
Positividad de Ki-67 en oat-cell en TMAs (HE
x10).

Fig. 11.
Positividad de Ki-67 en adenocarcinoma de pulmón en bloque (HE x20).
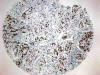
Fig. 12:
Positividad de Ki-67 en adenocarcinoma de pulmón
en TMAs (HE x10).
EGFR: La concordancia entre los cilindros TMAs y el bloque fue alta: en los Adenocarcinomas de Colon y en los Oat-Cell fue absoluta; en los Adenocarcinomas de pulmón se observó la concordancia del 83,3% del total y en los Carcinomas escamosos del 88,9%, porque aquí hubo cilindros no valorables y algunos perdidos durante el procesamiento (figs. 13-18).

Fig. 13:
Positividad de EGFR en adenocarcinoma de pulmón
en bloque (HE x10).
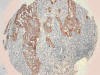
Fig. 14:
Positividad de EGFR en adenocarcinoma de pulmón
en TMAs (HE x10).
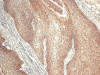
Fig. 15:
Positividad de EGFR en carcinoma escamoso en
bloque (HE x10).
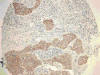
Fig. 16:
Positividad de EGFR en carcinoma escamoso en TMAs
(HE x10).

Fig. 17:
Positividad de EGFR en oat-cell en bloque (HE
x10).
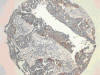
Fig. 18:
Positividad de EGFR en oat-cell en TMAs (HE x10).
Los diagnósticos de la muestra completa de 62 tumores dieron muy buenos resultados con los criterios no-paramétricos (tablas 1 y 2).
Los resultados del test Wilcoxon de los rangos en el Bloque demostró la posibilidad de concordancia en la tinción del marcador EGFR y el Ki-67 categorizado: entre 62 tumores hay 59 o 95,16% coincidencias en el grado de expresión categorizado de los dos marcadores; el resto de los casos no coincidió por falta de la medición en uno de los marcadores: en dos casos (3,22%, Adenocarcinomas de Colon) el bloque contenía las células insuficientes para Ki-67, sin embargo, EGFR fue bien definido; en un caso (1,61%, Oat Cell) no se realizó la prueba de EGFR, y sí la del Ki-67.
DISCUSIÓN
Al igual que en los diferentes estudios realizados para comprobar la utilidad de los TMAs, nosotros pudimos ver que los TMAs son representativos del tumor, tanto cuantitativamente como cualitativamente.
Aunque pudimos ver que existe un cierto porcentaje, entre un 20 y un 30%, donde no es tan válido la interpretación cuantitativa, esto sucedió principalmente en las áreas periféricas de los tumores, tanto en el bloque como los TMAs. También se vio que esta variación dependía del tipo de tumor.
Al realizar el análisis con la interpretación en bajo, moderado o alto grado de expresión, no hubo variaciones tan significativas, al igual que en el análisis de EGFR que hubo más del 90% de concordancia.
Por lo cual es importante tener en cuenta la heterogeneidad de los tumores, aunque se ha demostrado que no se altera la identificación de parámetros pronósticos.
Como hemos descrito, la utilización de esta técnica tiene grandes ventajas que hacen posible efectuar un estudio en un tiempo escaso. Ello se halla motivado por una rápida velocidad en el procesamiento de las muestras, la posibilidad de análisis simultáneo y estandarizado de múltiples muestras y la existencia de un área de tamaño suficiente para analizar. Nos damos cuenta que son una herramienta poderosa para un rápido screening tanto de marcadores como de alteraciones de diferentes tipos de tumores (9,21,22).
Pero no podemos olvidar que tiene ciertas limitaciones como el que es posible que en ocasiones un cilindro no tenga tejido tumoral o que el cilindro se pierda durante el proceso. Por lo cual, no lo recomiendan para estudio de nuevos antígenos, donde la distribución dentro del tejido no se conoce y se contraindica en el estudio de marcadores de expresión focal, ya que puede dar lugar a una interpretación errónea. En este caso, una alternativa sería la utilización de técnicas que requieran de un mayor aporte de material, como los «macrochop» (23).
Además, esta técnica también puede ser útil para el estudio de enfermedades no neoplásicas y utilizando citologías (24), aunque estas aplicaciones todavía se encuentran en una fase de estudio.
Tienen el potencial de acelerar estudios moleculares que busquen una asociación entre cambios moleculares y las características clinicopatológicas del tumor. Es importante saber que los TMA están diseñados para examinar tumores de una población y no tumores individuales. Puede ser utilizada como una herramienta de cribaje a nivel poblacional (22).
AGRADECIMIENTOS
Debemos un especial agradecimiento a Teresa Hernández Iglesias por la idea, diseño y elaboración del primer tissue array manual en el HUCA. Dos de los autores son miembros del IUOPA, financiado por la Obra Social Cajastur y por el programa de la RTICCC del ISCIII.
BIBLIOGRAFÍA
Wilson CA, Dering J. Recent translational research: microarray expression profiling of breast cancer-beyond classification and prognostic markers? Breast Cancer Res 2004; 6: 192-200.
Gillespie JW, Gannot G, Tangrea MA, Ahram M, Best CJ, Bichsel VE, Petricoin EF, Emmert-Buck MR, Chuaqui RF. Molecular profiling of cancer. Toxicol Pathol 2004; 32: 67-71.
Abdulkader I, Sanchez L, Cameselle-Teijeiro J, Gude F, Chavez JE, Lopez-Lopez R, Forteza J, Fraga M. Cell-cycle-associated markers and clinical outcome in human epithelial cancers: A tissue microarray study. Oncol Rep 2005; 14: 1527-31.
Misek DE, Imafuku Y, Hanash SM. Application of proteomic technologies to tumor analysis. Pharmacogenomics. 2004; 5: 1129-37.
Battifora H, Mehta P. The Checkerboard Tissue Block. An Improved Multitissue Control Block. Lab Invest 1990; 63: 722-4.
Bertucci F, Viens P, Tagett R, Nguyen C, Houlgatte R, Birnbaum D. DNA arrays in clinical oncology: promises and challenges. Lab Invest 2003; 83: 305-16.
Schraml P, Kononen J, Bubendorf L, Moch H. Tissue Microarrays for Gene Amplification Surveys in Many Different Tumor Types. Clin Cancer Res 1999; 5: 1966-75.
Kononen J, Bubendorf L, Kallioniemi A. Tissue Microarrays for High-throughput Molecular Profiling of Tumor Specimens. Nature Med 1998; 4: 844-7.
Simon R, Mirlacher M, Sauter G. Tissue microarrays. Methods Mol Med 2005; 114: 257-68.
Bubendorf L, Kononen J, Koivisto P. Survey of Gene Amplifications During Prostate Cancer Progression by High-throughput Fluorescence in situ Hybridization on Tissue Microarrays. Cancer Res 1999; 59: 803-6.
Fejzo MS, Slamon DJ. Frozen Tumor Tissue Microarray Technology for Analysis of Tumor RNA, DNA and Proteins. Am J Pathol 2001; 159: 1645-50.
Moch H, Kononen J, Kallioniemi O. Tissue Microarrays: What Will They Bring to Molecular and Anatomic Pathology? Adv Anat Pathol 2001; 8: 14-20.
Nohle D, Hackman BA, Ayers LW. The tissue micro-array data exchange specification: a web based experience browsing imported data. BMC Med Inform Decis Mak 2005; 5: 25.
Liu CL, Montgomery KD, Natkunam Y, West RB, Nielsen TO, Cheang MC, Turbin DA, Marinelli RJ, van de Rijn M, Higgins JP. TMA-Combiner, a simple software tool to permit analysis of replicate cores on tissue microarrays. Mod Pathol 2005; 18: 1641-8.
Camp RL, Charette LA, Rimm DL. Validation of Tissue Microarray Technology in Breast Carcinoma. Lab Invest 2000; 80: 1943-9.
Hoos A, Cordon-Cardo C. Tissue Microarray Profiling of Cancer Specimens and Cell Lines: Opportunities and Limitations. Lab Invest 2001; 81: 1331-8.
Nocito A, Kononen J, Kallioniemi OP. Tissue Microarrays for High-throughput Molecular Pathology Research. Int J Cancer 2001; 94: 1-5.
Rubin MA, Dunn R, Strawderman M. Tissue Microarray Sampling Strategy for Prostate Cancer Biomarker Analysis. Am J Surg Pathol 2002; 26: 312-9.
Gillett CE, Springall RJ, Barnes DM. Multiple Tissue Arrays in Histopathology Research: a validation study. J Pathol 2000, 192: 549-53.
Goldstein N, Armin M. Epidermal Growth Factor Receptor Inmunohistochemical Reactivity in Patients with American Joint Committe on Cancer Stage IV Colon Adenocarcinoma. Cancer 2001; 92: 1331-45.
Tzankov A, Went P, Zimpfer A, Dirnhofer S. Tissue microarray technology: principles, pitfalls and perspectives-lessons learned from hematological malignancies. Exp Gerontol 2005; 40: 737-44.
Al Kuraya K, Simon R, Sauter G. Tissue microarrays for high-throughput molecular pathology. Ann Saudi Med. 2004; 24: 169-74.
Fernández PL, Nayach I, Fernández E. Tissue Macroarrays («macrochops») for Gene Expression Analisis. Virchows Arch 2001; 438: 591-4.
Rimm DL. Impact of microarray technologies on cytopathology. Acta Cytol 2001; 45: 111-4.